前言
2023年,安序源与美国布朗大学Peter Belenky教授课题组(以下简称“课题组”)开展基于纳米孔基因测序技术的科研合作。
在顺利交付安序源AxiLona AXP-100平台后,课题组持续利用该平台进行微生物领域的测试。2024年7月,课题组在微生物组研究期刊Frontiers in Microbiomes上发表了研究论文。
16S rRNA全长1.5kb,传统二代测序因读长较短(50-150bp)仅能对部分区段设计引物扩增并测序。课题组基于测试数据,认为高精度的长读长测序可以轻松测通16S rRNA全长序列,提供更高分辨率的菌群分析能力,使得复杂样本(如肠道样本)的分析结果更简单、更可信。
概述
肠道微生物组的研究,尤其是其对于宿主的影响和互相作用、以及在不同环境暴露下的调节,一直是众多科研工作者关注的焦点。随着测序技术的成熟和发展,研究者能在种属水平对微生物种群进行深入的分析,从而探究不同因素影响下微生物的构成及其适应、进化的过程,以及对宿主造成的影响。
文章阐述了利用16S rRNA测序与生物信息学分析来探究饮食、性别及早期环境对阿莫西林暴露下小鼠微生物组的干扰及其恢复情况。研究团队在传统短读长测序方法外,对安序源AxiLona AXP-100长读长测序方法的结果进行了分析与比较,揭示了高精度长读长测序对微生物种群分析的应用潜力。

布朗大学Peter Belenky教授课题组与安序源应用支持团队
方法与结论
研究团队选取了Jackson Laboratory来源的雄性(MJ)和雌性 (FJ)C57BL/6 小鼠,以及Charles River Laboratories来源的雌性(FC)小鼠,分组进行高糖、高脂肪(西式)饮食和标准高纤维饲料(普通饲料)饮食喂养,通过对粪便样本进行16S rRNA 测序和生物信息学分析,研究微生物组对抗生素的反应。
通过测序与分析,研究团队指出,每组小鼠的微生物组构成在使用抗生素前就有不同的起始状态,这取决于供应商、性别和饮食。而抗生素的使用进一步加剧了这些差异,也表明每组小鼠对这种干扰的反应各不相同。研究团队发现,与使用抗生素的普通饲料组相比,西式饮食下的微生物组对抗生素的反应更为强烈,其 α 多样性、β 多样性以及微生物组成的变化更大。研究团队认为,这些发现强调了在研究抗生素对小鼠的影响时,考虑供应商来源、饮食和性别因素的重要性。同时指出,在开发人类治疗方法时,很可能需要采用基于患者特定微生物组信息的方法,以便在使用抗生素期间保护微生物组。
AxiLona AXP-100测序结果分析
在传统二代测序技术之外,研究团队使用安序源的AxiLona AXP-100高精度纳米孔测序技术测试了部分样本并对数据结果进行了分析,以确定抗生素作用于西式饮食组的FJ小鼠后,不同微生物菌种的丰度变化。
研究团队在第25天这个时间点上,从11个样本中得到了抗生素暴露组与未暴露组中丰度最高的前 30个科的微生物群落数据。在分析第 25 天这个时间点的3个暴露组样本和1个未暴露组样本时,研究团队观察到在抗生素暴露条件下,肠杆菌科和阿克曼氏菌科的数量有所增加。在阿克曼氏菌科中,共观察到两个菌种,即嗜黏蛋白阿克曼氏菌(Akkermansia muciniphila)和嗜糖阿克曼氏菌(Akkermansia glycaniphila)(图 5B)。团队发现,经过抗生素处理后,嗜糖阿克曼氏菌数量减少,而嗜黏蛋白阿克曼氏菌数量增加。在肠杆菌科的63个观察到的菌种中,一些数量最多的菌种包括大肠杆菌、密歇根克雷伯菌、产酸克雷伯菌和类肺炎克雷伯菌(图 5C)。
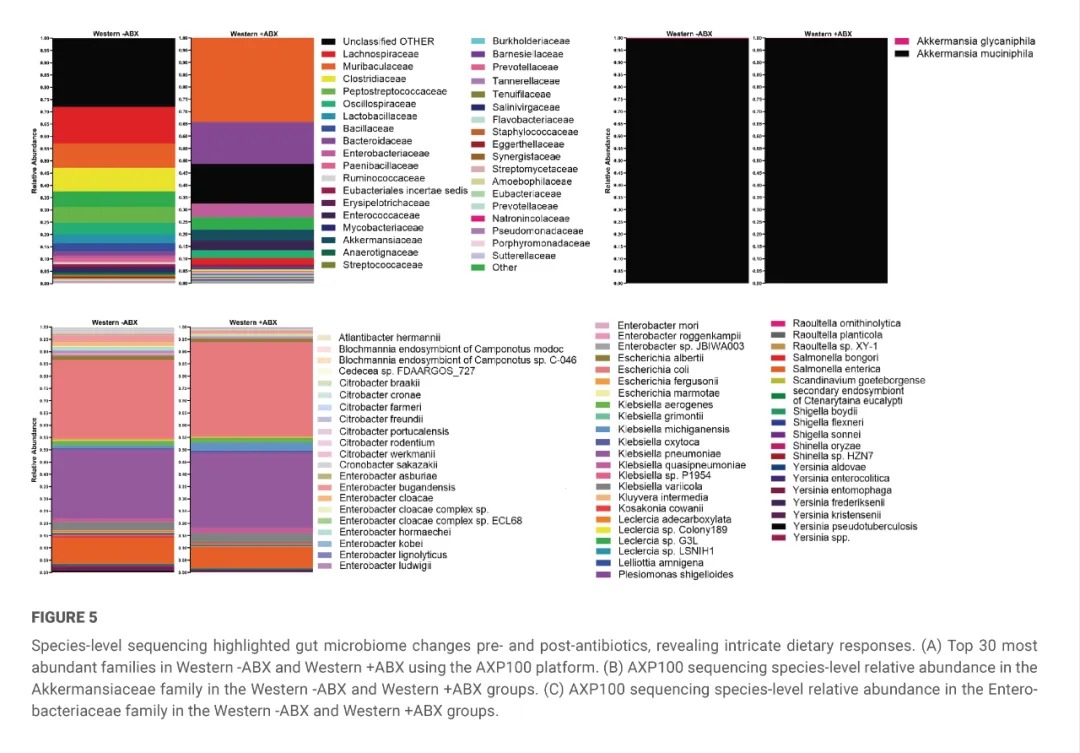
研究团队认为,尽管这项分析的样本量(n)不足以开展统计分析,但它确实说明了AxiLona AXP-100长读长测序技术可用于开展微生物菌种层面的分析。

布朗大学Peter Belenky教授与安序源应用副总裁Nikolay Sergeev博士

